长链非编码RNA ITGB-1对甲状腺乳头状癌细胞侵袭、迁徙与增殖能力的影响
摘要:目的:探究长链非编码RNA(long non-coding RNA,lncRNA) ITGB-1在甲状腺乳头状癌(papillary thyroid carcinoma,PTC)中的表达水平及对PTC细胞侵袭、迁徙与增殖能力的影响。方法:收集34例2018年9月~2020年1月于西南医科大学附属医院甲状腺外科手术切除的PTC标本以及对应的癌旁正常组织,采用实时荧光定量逆转录聚合酶链反应(quantitative real-time polymerase chain reaction,qRT-PCR)检测样本中lncRNA ITGB-1的表达情况并分析临床病理的相关性。通过qRT-PCR检测ITGB-1在PTC细胞TPC-1和正常甲状腺细胞Nthy-ori3-1的表达水平。通过transwell实验、划痕实验和CCK8实验分别探究siRNA技术下调ITGB-1对侵袭、迁徙和增殖能力的影响。结果:LncRNA ITGB-1在PTC癌组织中的表达水平高于癌旁正常组织,并且在PTC癌组织中的表达水平与淋巴结转移、被膜及腺体外侵犯有关。ITGB-1在PTC细胞TPC-1中的表达高于正常甲状腺细胞Nthy-ori3-1。下调ITGB-1表达后,TPC-1的侵袭、迁徙和增殖能力均受到明显抑制。结论:LncRNA ITGB-1在PTC中表达增高且与其发生发展相关,下调ITGB-1表达可抑制PTC细胞TPC-1的侵袭、迁徙与增殖能力。
关键词:甲状腺乳头状癌; lncRNA ITGB-1; 侵袭; 迁徙; 增殖;
甲状腺癌是头颈部最常见的内分泌恶性肿瘤,甲状腺乳头状癌(papillary thyroid carcinoma,PTC)是其最常见的病理类型。研究发现,PTC的发病率正呈逐年上升的趋势[1]。长链非编码RNA(long non-coding RNA,lncRNA)由200多个核苷酸残基组成,研究表明lncRNA参与了X染色体沉默、基因组印记以及染色质修饰、转录干扰、转录激活、核内运输等多种重要的调控过程,并且lncRNA与肿瘤的关联也越发引人关注[2-3]。ITGB-1是lncRNA家族中的一员,目前研究者们已经在多种癌症中发现其与癌细胞的侵袭、迁徙及增殖能力相关,但在PTC领域尚未见明确报道[4]。本文以PTC癌组织及癌旁正常组织、TPC-1细胞为研究基础,探讨ITGB-1在PTC中的表达情况,并分析下调ITGB-1对TPC-1细胞侵袭、迁徙及增殖能力的影响。
1 材料与方法
1.1 材料
1.1.1 组织样本
34例患者的组织标本来源于2018年9月~2020年1月在西南医科大学附属医院甲状腺外科接受手术切除的PTC患者的癌组织以及对应的癌旁正常组织(定义为与肿瘤的距离大于1cm以上)。本实验纳入的所有患者均由2位本院病理科医师明确诊断为PTC,所有患者术前均未接受其他治疗。34例患者年龄在20~73岁之间,平均年龄为(41.88±12.97)岁;男性12例,女性22例,其中8例患者年龄大于55岁,20例有淋巴结转移,6例患者存在多发癌灶,17例患者属于微小乳头状癌,11例见被膜或腺体外侵犯。参考美国癌症联合会指南第8版,Ⅰ期患者30例,Ⅲ期和Ⅳ期患者各2例。本实验经西南医科大学附属医院伦理委员会批准,所有患者均签署知情同意书。
1.1.2 细胞株及来源
人甲状腺乳头状细胞癌细胞株TPC-1和人正常甲状腺滤泡上皮细胞株Nthyori3-1均来自中国科学院干细胞库。
1.1.3 主要试剂及来源
本研究使用的试剂和材料有:RPMI培养基1640(Hyclone);Gibco胎牛血清、F12K培养基(Thermo Fisher);RT试剂盒Rever Tra Ace q PCR RT Kit(TOYOBO);Quanti Nova SYBR Green PCR Kit购(Qiagen);细胞、动物RNA提取试剂盒(成都福际生物公司);siRNA序列、阴性对照序列、转染试剂(广州锐博公司);lncRNA ITGB-1序列、内参GAPDH序列(上海生工生物工程公司);细胞消化胰酶、结晶紫溶液、组织固定液、CCK8(Cell Counting Kit-8)试剂盒(上海碧云天生物技术公司)。
1.2 方法
1.2.1 组织样本
所有新鲜组织均于液氮中速冻24 h后,放入-80℃冰箱储存。
1.2.2 组织样本RNA提取、逆转录及实时荧光定
量逆转录聚合酶链反应(quantitative real-time polymerase chain reaction,qRT-PCR)采用动物RNA提取试剂盒分别提取PTC组织和癌旁正常组织的总RNA,使用分光光度计检测总RNA浓度及纯度(A260/A280在1.8~2.0,RNA浓度在300~500 ng之间),琼脂凝胶电泳成像检测RNA完整性;采用逆转录试剂盒对所提取的RNA进行逆转录得到cDNA。利用QuantiNova SYBR Green PCR Kit将所得到的cDNA及目的基因ITGB-1、内参GAPHDH配成实时荧光定量的反应体系后,于qRT-PCR仪上检测。所涉及的引物序列如下:ITGB-1上游引物:5’-CCTCTCAGCCTCCAGCGTTG-3’,下游引物:5’-TGCTCTTGCTCACTCACACTCC-3’;GAPDH上游引物:5’-CAAATGACCCCTTCATTGACC-3’,GAPDH下游引物:5’-GACAAGCTTCCCGTTCTCAG-3’。
1.2.3 细胞培养
TPC-1细胞采用10%胎牛血清的RPMI 1640培养基进行培养,置于5%CO2、37℃恒温培养箱;Nthy-ori3-1细胞采用10%胎牛血清的F12K培养基进行培养,置于5%CO2、37℃恒温培养箱。
1.2.4 细胞RNA提取、逆转录及qRT-PCR
采用细胞RNA提取试剂盒分别提取TPC-1细胞、Nthyori3-1细胞的总RNA,余步骤同上述组织提取部分。
1.2.5细胞分组及培养试剂差异
以TPC-1细胞设置空白对照组、阴性对照组和目的基因组,于空白对照组加入常规细胞培养基,阴性对照组加入含有无意义序列的siRNA的转染体系培养基,目的基因组加入含siRNA序列的转染体系培养基,每组均3个副孔。
1.2.6 转染效率测定
消化培养瓶中均匀生长且细胞密度大约为80%的TPC-1细胞后均匀种植于6孔板中,待细胞完全贴壁且密度大约为每孔面积50%左右进行转染,根据分组配置培养基,培养24 h提取细胞RNA,并逆转录为cDNA后行qRT-PCR检测。所涉及的基因序列如下:siRNA靶序列为5’-GCAGCTGTTTCCAGAATATTGC-3’,si-Ctrl的靶序列5’-UUCUCCGAACGUGUCACGUTT-3’。
1.2.7 Transwell实验检测侵袭能力
消化培养瓶中均匀生长且细胞密度大约为80%的TPC-1细胞后均匀种植于24孔板中,待细胞完全贴壁且每孔密度约50%左右进行转染,根据分组配置培养基,培养24 h后经消化、终止消化、洗涤、离心后,于每个transwell小室内接种40 000个细胞,内层加入无血清的培养基,外层加入含10%胎牛血清的培养基,继续培养24 h后取出,磷酸盐缓冲液(phosphate buffer saline,PBS)清洗小室内外,棉签轻柔擦拭小室内层,重复3次,用组织固定液固定细胞10 min,0.1%的结晶紫染色10 min,PBS漂洗、晾干后于显微镜下拍照并计数穿过小室的细胞个数。
1.2.8 划痕实验检测迁徙能力
消化培养瓶中均匀生长且细胞密度大约为80%的TPC-1细胞后均匀种植于已标记观察点的12孔板中,待细胞完全贴壁且每孔密度约50%左右进行转染,根据分组配置培养基,培养24 h后,用20μL加样枪尖在每个细胞培养孔内划痕(作为0 h);磷酸盐缓冲液小心洗涤细胞三次清除划下的细胞,加入无血清的培养基,继续培养细胞24 h;在显微镜下观察细胞的划痕愈合能力,计算划痕愈合率,公式:(0 h划痕距离-24 h划痕距离)/0 h划痕距离。
1.2.9 CCK8实验检测增殖能力
消化培养瓶中均匀生长且细胞密度大约为80%的TPC-1细胞后均匀种植于24孔板中,待细胞完全贴壁且每孔密度约50%左右进行转染,根据分组配置培养基,培养24 h后经消化、终止消化后接种于96孔板中,每孔细胞数约3 000个,并于24 h、48 h、72 h分别向孔中加入10μL CCK溶液和100μL无血清培养基混合液,37℃避光孵育1.5 h后检测吸光度(optical density,OD值)。
1.3 统计学及图片处理
用SPSS 23.0对数据进行统计分析,S-W检验测试数据是否符合正态分布,如果数据符合正态分布,两组间运用t检验,多组间采用单因素方差分析;如数据不符合正态分布,则使用非参数检验,P<0.05为差异有统计学意义(*:P<0.05,**:P<0.01,***:P<0.001)。Graph PAD 8.0、Image J软件辅助做图。
2 结果
2.1 ITGB-1在PTC组织和癌旁正常组织中的表达情况
通过qRT-PCR实验分析,我们发现ITGB-1在PTC组织中的表达水平高于癌旁正常组织,差异有统计学意义(P<0.05)(图1)。

图1 ITGB-1在PTC组织和癌旁正常组织中的表达差异(*:P<0.05)
Figure 1.Relative Expression of ITGB-1 in PTC Tissues and Adjacent Normal Tissues(*:P<0.05)PTC:Papillary thyroid carcinoma.
2.2 ITGB-1的表达与PTC组织的临床病理相关性
与无淋巴结转移的患者相比,有淋巴结转移的患者癌组织中ITGB-1的表达显著更高,差异有统计学意义(P<0.001);与无被膜或腺体外侵犯的患者相比,有被膜或腺体外侵犯的患者癌组织中ITGB-1的表达水平也显著更高,差异有统计学意义(P<0.01)。另外,ITGB-1的表达与患者性别、年龄、肿瘤大小、是否为多灶癌无确切关系(表1)。
表1 临床病理特征与甲状腺乳头状癌中ITGB-1的表达关系
2.3 ITGB-1在TPC-1细胞和Nthy-ori3-1细胞中的表达情况
通过qRT-PCR实验分析,我们发现ITGB-1在TPC-1细胞中的表达水平高于Nthy-ori3-1细胞,差异有统计学意义(P<0.05)(图2)。
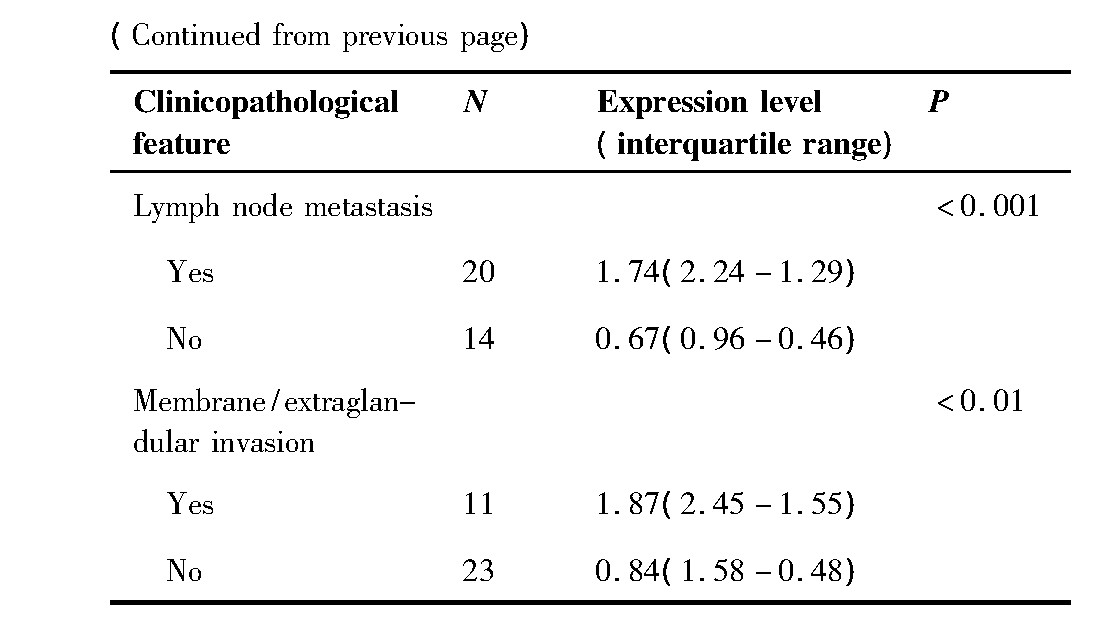
图2 ITGB-1在TPC-1和Nthy-ori3-1细胞中的表达差异(*:P<0.05)
Figure 2.Relative Expression of ITGB-1 in TPC-1 and Nt-hy-ori3-1 Cells(*:P<0.05)
2.4 转染效率的测定
通过qRT-PCR实验我们发现,加入无意义序列的siRNA的阴性对照组相比,加入针对ITGB-1序列的siRNA的目的基因组中ITGB-1的表达明显降低,差异有统计学意义(P<0.01),故siRNA可促使IT-GB-1在TPC-1中表达降低(图3)。
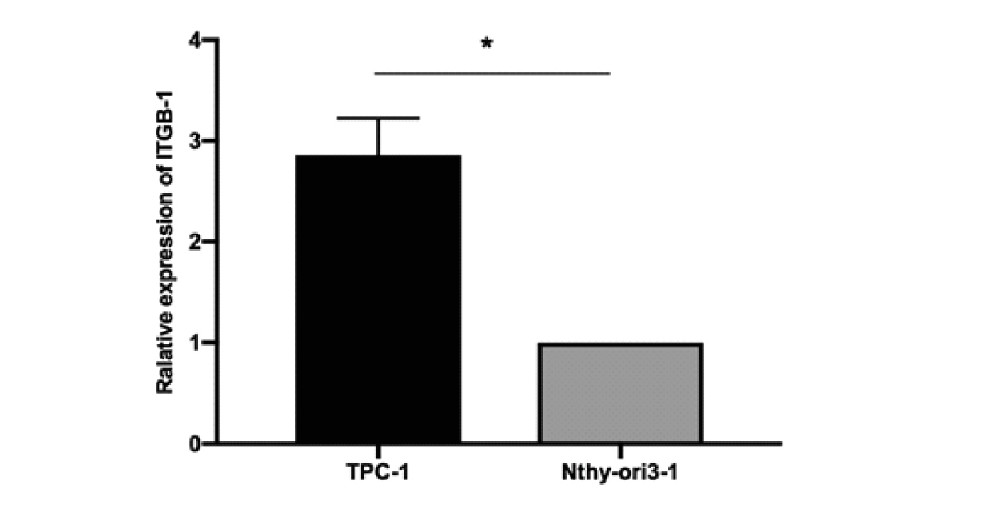
图3 检测siRNA的转染效率(**:P<0.01)
Figure 3.Ralative Expression of ITGB-1 in Target Gene Group and Negative Control Group(**:P<0.01)
2.5 Transwell检测细胞侵袭能力
目的基因组、阴性对照组、空白对照组中,TPC-1细胞个数分别为(171.33±20.79)个、(471.33±24.09)个、(514.67±41.79)个。目的基因组与阴性对照组的差异有统计学意义(P<0.001),目的基因组与空白对照组的差异亦有统计学意义(P<0.001),而阴性对照组与空白对照组的差异则无统计学意义(P=0.124),故下调ITGB-1可抑制TPC-1细胞的侵袭能力(图4)。
图4 Transwell实验三组细胞数目差异(结晶紫染色,40×10)
Figure 4.Number of Cells in Three Groups in Transwell Migration Assay(Crystal Violet Staining,40×10)
2.6 划痕实验检测细胞迁徙能力
在目的基因组、阴性对照组、空白对照组中,TPC-1细胞24 h的迁徙率分别为26.01%±2.29%、40.40%±2.88%、40.60%±4.07%。目的基因组与阴性对照组的差异有统计学意义(P<0.01),目的基因组与空白对照组的差异亦有统计学意义(P<0.01),而阴性对照组与空白对照组的差异则无统计学意义(P=0.942),故下调ITGB-1可抑制TPC-1细胞的迁徙能力(图5)。

图5 划痕实验中三组细胞迁徙率的差异(**:P<0.01)
Figure 5.Migration Rate of TPC-1 Cells in Three Groups in Scratch Assay(**:P<0.01)
2.7 CCK8实验检测细胞增殖能力
在目的基因组、阴性对照组、空白对照组中,TPC-1细胞24 h的OD值分别为0.30±0.01、0.50±0.05、0.63±0.05;48 h的OD值分别为0.71±0.02、0.97±0.04、1.05±0.01;72h的OD值分别为1.36±0.06、2.04±0.21、2.02±0.01。24 h时,目的基因组与阴性对照组的差异有统计学意义(P<0.05),目的基因组与空白对照组的差异亦有统计学意义(P<0.05),而阴性对照组与空白对照组的差异则无统计学意义(P=0.127);48 h时,目的基因组与阴性对照组的差异有统计学意义(P<0.05),目的基因组与空白对照组的差异亦有统计学意义(P<0.01),而阴性对照组与空白对照组的差异则无统计学意义(P=0.126);72 h时,目的基因组与阴性对照组的差异有统计学意义(P<0.05),目的基因组与空白对照组的差异亦有统计学意义(P<0.01),而阴性对照组与空白对照组的差异则无统计学意义(P=0.925)。可见,下调IT-GB-1可抑制TPC-1细胞的增殖能力(图6)。

图6 CCK8实验中3组在24、48、72 h下的OD值差异
Figure 6.OD450 Values in Three Groups at 24,48,and72 Hours in CCK8 Assay
3 讨论
近年来,随着医疗技术的发展及人类健康意识的增强,甲状腺癌发病检出率逐年递增,有报告指出其可能成为全球第四大癌症[5],并且呈现年轻化趋势[6]。PTC是最常见的甲状腺癌类型,约占85%~90%[7]。根据研究报道,30%~70%的PTC患者会出现颈部淋巴结转移,且预后相对较差[8];而癌灶突破甲状腺包膜生长或形成腺体外侵犯,其预后也相对较差[9-10]。故而对PTC侵袭、迁移和增殖能力的调控就颇显重要。
目前分子标记物已被广泛用于多种疾病的临床诊断和预后判断过程。作为近几年的肿瘤与分子生物学的研究热点[11-12],越来越多研究表明lncRNA与PTC的发生发展密切相关[13]。比如,Xia等[14]发现CCND2-AS1促进PTC的增殖,迁移和侵袭;Wang等[15]发现PTCSC3通过Wnt/β-catenin信号影响PTC的增殖和迁移;Zhang等[16]发现RGMB-AS1被E2F1激活并促进PTC增殖和侵袭。与此同时,IT-GB-1已经被验证在诸多癌症的发展过程中起一定作用。例如,Huang等[17]发现ITGB-1可通过上调ROCK1促进人肝癌细胞增殖,迁移和侵袭;Shang等[18]发现ITGB-1能促进人肝癌细胞的细胞增殖和迁移;Dai等[19]发现ITGB-1可通过调节microRNA-10a表达促进膀胱癌的发展;Zheng等[20]发现ITGB-1通过下调Mcl-1促进透明细胞肾细胞癌的迁移和侵袭。
本研究通过检测34对PTC组织及相应的癌旁正常组织标本中ITGB-1的表达情况,发现ITGB-1在PTC组织中呈现相对较高的表达水平,提示ITGB-1或许参与了PTC的发生发展,这种差异性可为今后PTC与正常甲状腺组织的初步鉴别提供另一种可能性。局部侵犯及远处转移是恶性肿瘤常见的特征。PTC虽然被公认为“惰性”的恶性肿瘤,常常表现出轻度的临床行为及良好的预后,但目前手术切除仍旧是治疗PTC的主要手段[21]。病灶突破甲状腺被膜向腺体外生长,可能导致周围结构受侵,如嵌入气管、食道,包绕颈部大血管,累及喉返神经、甲状旁腺等,常常增加手术难度,甚至无法全部切除;而术前已经存在局部转移的淋巴结,往往需要面临扩大手术范围及存在术中未切净转移灶导致术后易出现复发可能;术前评估发现远处转移,如肺、脑、骨等情况,至今尚无公认的有效治疗方案。目前很多专家学者将淋巴结转移及被膜或腺体外侵犯视为评估PTC的独立危险因素。术前良好的评判有无淋巴结转移或被膜及腺体外侵犯能够更好行术前准备,提高手术成功率。术后利用有效的手段对存在淋巴结转移或被膜及腺体外侵犯的患者的监控能够及时评估复发转移的出现。结合临床病理分析,ITGB-1在有淋巴结转移或存在被膜及腺体外侵犯的PTC患者中的表达显著高于无淋巴结转移或不存在被膜及腺体外侵犯的PTC患者。ITGB-1可能可以作为示警淋巴结转移或被膜及腺体外侵犯的PTC的分子标记物。目前对于一些常见的、恶性程度高、临床分期较晚的恶性肿瘤,在细胞分子水平上,针对已经明确的致癌位点进行特异性的靶向治疗已经逐渐增多。虽然由于PTC被视“惰性”肿瘤,且远处转移发生率极少,靶向治疗的研究尚有欠缺,但精确化的靶向治疗仍旧是目前及未来所需要的治疗手段。本研究依托TPC-1细胞为基础,在细胞学功能实验中,我们发现通过下调ITGB-1的表达可以显著抑制TPC-1细胞的侵袭、迁徙和增殖能力。这提示ITGB-1在PTC细胞层面抑制了其生长、运动。我们推测通过下调ITGB-1的基因治疗手段,也许未来可能成为PTC靶向治疗的一项潜在选择。
综上所述,本研究通过临床与基础实验的结合,初步探讨了ITGB-1在PTC组织和TPC-1细胞中的作用,结合结果推测ITGB-1能很可能在PTC的发生发展中起着正性调控,且ITGB-1对PTC细胞的迁移、增殖能力具有促进效应。其更进一步作用机制是我们下一步的研究重点。
参考文献
[1]Siegel RL,Miller KD,Jemal A.Cancer statistics,2016[J].Ca ACancer J Clin,2016,66(1)∶7-30.
[2]Pan YF,Feng L,Zhang XQ,et al.Role of long non-coding RNAs in gene regulation and oncogenesis[J].Chin Med J(Engl),2011,124(15)∶2378-2383.
[3]Maass PG,Luft FC,Bhring S.Long non-coding RNA in health and disease[J].J Mol Med(Berl),2014,92(4)∶337-346.
[4]Yan M,Zhang LN,Li GQ,et al.Long noncoding RNA linc-ITGB1promotes cell migration and invasion in human breast cancer[J].Biotechnol Appl Biochem,2017,64(1)∶5-13.
[5]Kim J,Gosnell JE,Roman SA.Geographic influences in the global rise of thyroid cancer[J].Nat Rev Endocrinol,2020,16(1)∶17-29.
[6]任艳军,刘庆敏,葛明华,等.2010-2014年浙江省肿瘤登记地区甲状腺癌发病和死亡情况分析[J].中华预防医学杂志,2019,53(10)∶1062-1065.
[7]Grant CS.Papillary thyroid cancer:Strategies for optimal individualized surgical management[J].Clin Ther,2014,36(7)∶1117-1126.
[8]Shah JP.Re:Extent of surgery affects papillary thyroid cancer[J].Ann Surg,2008,247(6)∶1082-1083.
[9]Wang CY,Chen XH,Wei XY,et al.Recurrence factors and prevention of complications of pediatric differentiated thyroid cancer[J].Asian J Surg,2017,40(1)∶55-60.
[10]黄璐,李超,王薇,等.甲状腺癌腺外侵犯的意义及最新研究进展[J].中华耳鼻咽喉头颈外科杂志,2019,54(9)∶717-720.
[11]Lv XR,Li Y,Li YH,et al.FAL1:A critical oncogenic long noncoding RNA in human cancers[J].Life Sci,2019,236∶116918.
[12]Vance J,Gilani SM.Thyroid cytopathology:Updates and molecular testing[J].Pathologica,2019,111(2)∶51-57.
[13]Wang J,Zhang X,Chen W,et al.Regulatory roles of long noncoding RNAs implicated in cancer hallmarks[J].Int J Cancer,2020,146(4)∶906-916.
[14]Xia E,Bhandari A,Shen YY,et al.LncRNA CCND2-AS1 promotes proliferation,migration,and invasion in papillary thyroid carcinoma[J].Biochem Biophys Res Commun,2018,496(2)∶628-632.
[15]Wang XM,Lu XB,Geng ZS,et al.LncRNA PTCSC3/miR-574-5p governs cell proliferation and migration of papillary thyroid carcinoma via Wnt/β-Catenin signaling[J].J Cell Biochem,2017,118(12)∶4745-4752.
[16]Zhang Z,Li SY,Zhang LB.LncRNA RGMB-AS1 is activated by E2F1 and promotes cell proliferation and invasion in papillary thyroid carcinoma[J].Eur Rev Med Pharmacol Sci,2018,22(7)∶1979-1986.
[17]Huang L,Li XY,Gao WJ.Long non-coding RNA linc-ITGB1 promotes cell proliferation,migration,and invasion in human hepatoma carcinoma by up-regulating ROCK1[J].Biosci Rep,2018,38(5)∶BSR20181289.
[18]Shang ML,Xu XH,Zhang M.Long non-coding RNA linc-ITGB1promotes cell proliferation and migration in human hepatocellular carcinoma cells[J].Exp Ther Med,2017,14(5)∶4687-4692.
[19]Dai L,Chai CM,Shen TY,et al.LncRNA ITGB1 promotes the development of bladder cancer through regulating microRNA-10a expression[J].Eur Rev Med Pharmacol Sci,2019,23(16)∶6858-6867.
[20]Zheng XL,Zhang YY,Lv WG.Long noncoding RNA ITGB1 promotes migration and invasion of clear cell renal cell carcinoma by downregulating Mcl-1[J].Eur Rev Med Pharmacol Sci,2019,23(5)∶1996-2002.
[21]李超,蔡永聪,孙荣昊.甲状腺微小乳头状癌:选择手术还是非手术主动监测?[J].肿瘤预防与治疗,2019,32(12)∶1045-1050.






